Einführung in die Statistik
Vorwort mit Hinweisen zur Notation
R ist zweierlei: eine Programmiersprache und eine Open-Source-Software, die unter den Bedingungen der GNU General Public License (GPL) lizensiert wird – und daher in jedweder Weise frei genutzt, bearbeitet und weiterverbreitet werden darf. Sie zeichnet sich insbesondere für statistische Fragestellungen, für die Datenanalyse und -visualisierung aus. Ein kontinuierlich gepflegter Fundus an Zusatzpaketen, die über das Comprehensive R Archive Network (CRAN) installiert werden können, ermöglicht es, einfache wie schwere Probleme zu lösen, ohne entsprechende Funktionen selbst erstellen zu müssen. R ist aufgrunddessen sowohl in der Lage, simple arithmetische Berechnungen (Addition, Subtraktion, Multiplikation, Division) auszuführen, als auch komplexere Untersuchungen (Regressionsmodelle, Clusteranalyse) anzustoßen, die ohne Computerunterstützung zeitintensiv sind.
Ziele und Aufbau
Die Lehrmodule sollen Ihnen einen Überblick über die Möglichkeiten von R mit konkreten, für Geisteswissenschaftler relevanten Anwendungsbeispielen liefern und Sie an die Hand nehmen, erste eigene Analysen auszuführen – und vor allem zu meistern. Zunächst wird dafür die integrierte Entwicklungsumgebung RStudio eingeführt. Es folgen Grundlagen der Programmierung, welche die unterschiedlichen Datentypen und -strukturen in R erläutern und beispielhaft deren Unterschiede aufzeigen. Anschließend stehen Befehle zum Import und Export von Daten im Fokus. Im Punkt Datenaufbereitung werden daraufhin die Grundlagen angewandt und importierte Datensätze für die weitere statistische Auswertung vorbereitet. Es sind keinerlei informatische oder statistische Vorkenntnisse vonnöten – wohl aber das Interesse, über Datensätzen zu sitzen und zu knobeln.1 Auch wenn dies erst einmal mühselig klingen mag: Erste Erfolgserlebnisse zeigen sich mit R erstaunlich schnell.
Referenzen
Für die weiterführende Lektüre ist zum einen das offizielle Benutzerhandbuch An Introduction to R empfehlenswert, zum anderen der Bereich Frequently Asked Questions on R; beide sind in R Project verankert. Eine aktive Plattform, um Probleme zu diskutieren und Lösungen auszutauschen, findet sich zudem in dem Internetforum Stack Overflow, das Beiträge, die sich thematisch auf R beziehen, explizit als solche kennzeichnet. Nach einer entsprechenden Registrierung können dort auch eigene Fragen eingestellt und von anderen Mitgliedern beantwortet werden. Informationen zu den internen Hilfeseiten in R erhalten Sie hier im Abschnitt Entwicklungsumgebung RStudio -> Die interne Hilfefunktion.
Hinweise zur Notation
Alle Codebeispiele in den Modulen sind entsprechend gekennzeichnet
und können einfach kopiert und in die Konsole von R oder RStudio
eingefügt werden. Die Struktur wird an folgendem Minimalbeispiel
deutlich:
1+1 # Addition
Die erste Zeile gibt den vom System auszuführenden Code an
(
## [1] 21+1). Die Raute (#) kennzeichnet den Beginn
eines Kommentars. Alle Zeichen, die nach einer Raute in einer Zeile
stehen, werden von R nicht interpretiert – sie sind sozusagen kleinere
oder größere Klebezettel, die den vorhergehenden Code dokumentieren oder
mit Anmerkungen versehen (hier der Text Addition). Die
letzte Zeile zeigt den Output, also das Ergebnis, das entsteht,
wenn der vorhergehende Code ausgeführt wurde. Die zwei Rauten
(##) dienen einzig der visuellen Abgrenzung zu einem
Kommentar.
Anmerkungen
- Datenwissenschaftler bezeichnen sich nicht umsonst auch gerne als Data Wizards oder Data Monkeys.↩︎
Die Entwicklungsumgebung RStudio
Um den Einstieg in die Programmierung mit R zu erleichtern, bietet
sich die Verwendung einer integrierten Entwicklungsumgebung
(Integrated Development Environment, IDE) an. Solche
Umgebungen kombinieren mehrere Vorzüge: Sie interpretieren den Code
nicht nur, sodass ein Output erzeugt werden kann, sondern
erweitern ihre Funktionalität auch visuell, um die Arbeit produktiver zu
gestalten. Eine IDE zeigt beispielsweise an, wenn ein Objekt,
auf das referenziert wird, noch nicht existiert oder eine geschweifte
Klammer fehlt, die dazu dient, eine Funktion zu beenden. Ebenso können
Grafiken direkt im momentan aktiven Bildschirmfenster angezeigt werden,
ohne sie im angegebenen Zielverzeichnis erst suchen und dann öffnen zu
müssen. Ein beliebtes Instrument dieser Art für R ist RStudio, das Sie in
Ihrem Desktop
unter Applications -> Development finden. Bei Aufruf von
RStudio muss R nicht zusätzlich geladen werden.
Benutzeroberfläche
Im Gegensatz zu einer weiteren statistischen Software, SPSS, kommt R standartmäßig ohne grafische Benutzeroberfläche (Graphical User Interface, GUI) aus – Benutzer interagieren stattdessen über eine Kommandozeile (siehe Abbildung 1).
RStudio erweitert diese Funktionalität über Panels, die das Bildschirmfenster in vier Bereiche abtrennen und alle benötigten Ressourcen präsentieren. Zunächst sind beim ersten Aufruf jedoch nur drei Panels aktiv (Abbildung 2): Console (links), Workspace (rechts oben), Working Directory (rechts unten). Workspace und Working Directory enthalten mehrere Registerkarten, die eine weitere Untergliederung der Bereiche erlauben.
Die Funktionen und Inhalte der Panels seien im Folgenden kurz erläutert.
- Console
Das wichtigste Werkzeug in RStudio ist die Konsole – über sie werden Variablen unter anderem deklariert und manipuliert, Daten importiert und exportiert, Grafiken generiert und modifiziert. Kurzum: Jeder Befehl in RStudio läuft letztlich über die Konsole und stößt im Prozessor des Computers eine Aktion an. Wenn diese Aktion, beispielsweise die Addition zweier Zahlen, beendet wurde, schickt der Prozessor das Ergebnis an den Nutzer – und es erscheint als Output in der Konsole. Das Symbol>kennzeichnet wie in R die Eingabeaufforderung (auch Prompt). Diese Markierung verweist auf die Stelle, an der Befehle eingegeben werden können. Bereits ausgeführte Befehle wandern eine Zeile nach oben; die Eingabeaufforderung steht weiterhin fest in der letzten, also untersten Zeile der Konsole. - Workspace
Alle erzeugten Daten, Werte und Funktionen, ihre Dimension, Länge und Rückgabewerte (dazu mehr hier im Abschnitt Grundlagen in R) landen in der Registerkarte Environment im Panel Workspace. Im Reiter History finden Sie eine Übersicht der bisher von Ihnen in dieser Sitzung ausgeführten Befehle – also jede Codezeile, die seit dem Öffnen von RStudio über die Konsole bestätigt wurde. Eine hilfreiche Funktion: Bei Doppelklick auf einen Befehl in der History kopiert RStudio den entsprechenden Code in die Konsole, wo er nochmals aufgerufen werden kann. - Working Directory
Das Panel Working Directory hält weitere Übersichten parat: Files, Plots, Packages und Help. Files zeigt die Ordner- und Dateistruktur des gewählten Zielverzeichnisses, Plots alle in dieser Sitzung generierten Grafiken, Packages alle bislang im System installierten Pakete, Help die internen Hilfeseiten.
Einen ausführlichen Spickzettel mit weiteren Symbolerklärungen und einer Liste von Tastenkombinationen (sogenannten Shortcuts 1) stellt RStudio online bereit.
Erstellen und Speichern eines Skripts
Womöglich fragen Sie sich mittlerweile: Wo landen meine eingegebenen Befehle – und was passiert mit ihnen, wenn ich RStudio schließe? Damit Ihre Arbeit nicht in die ewigen virtuellen Jagdgründe eingeht, empfiehlt es sich, Code und Kommentare in ein Skript zu schreiben. Um ein neues Skript zu erstellen, gibt es drei Möglichkeiten:
- die Tastenkombination
[Strg] + [Umschalt] + [N]2, - den Menüeintrag
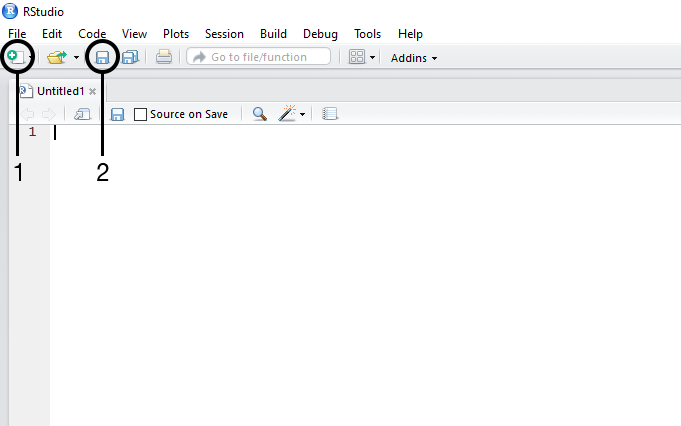
R Script, der überFile -> New Filezu erreichen ist, - das erste Symbol der obersten Symbolleiste: ein weißes Plus in einem
grünen Kreis, das auf einem weißen Quadrat steht (gekennzeichnet mit 1
in Abbildung
3). Wenn Sie auf das Symbol klicken, öffnet sich ein
Dropdown-Menü. Wählen Sie dort
R Scriptan, damit ein neues Skript erstellt wird.
Ein Skript ist im Grunde ein gewöhnliches Textdokument – nur handelt es sich bei Ihrem Text eben um eine Sammlung aus Befehlen, die untereinander stehen und in RStudio aufgerufen werden können. Mit Erstellen eines Skripts erhalten Sie auch das letzte, vierte Panel, das die Konsole nun im Bildschirmfenster nach links unten rückt und darüber Platz nimmt (Abbildung 4).
Eine Empfehlung: Arbeiten Sie nur direkt über die Konsole, wenn es
sich um Code handelt, der nicht maßgeblich für Ihre Auswertung ist und
später keiner erneuten Ausführung bedarf (beispielsweise der Aufruf
einer internen Hilfeseite). Im Zweifel gilt jedoch: Schreiben Sie jeden
Befehl in Ihr Skript und rufen Sie den jeweiligen Befehl auch über das
Skript auf (wie dies funktioniert, lesen Sie im Abschnitt Ausführen von Code).
Auch für das Speichern Ihres Skripts gibt es mehrere
Möglichkeiten:
- die Tastenkombination
[Strg] + [S], - den Menüeintrag
Save, der überFilezu erreichen ist, - das Diskettensymbol der obersten Symbolleiste (gekennzeichnet mit 2 in Abbildung 3).
Es öffnet sich ein Dialogfenster, über das Sie auswählen können, in
welchem Verzeichnis Ihr Skript gespeichert werden soll. Der Dateiname
muss nicht die Endung .R beinhalten – diese wird
automatisch ergänzt. Falls beim Speichern nach Encoding gefragt
wird, wählen Sie ISO-8859-1, um deutsche Umlaute darstellen
zu können.
Ausführen von Code
Mittlerweile liegt ein jungfräuliches Skript vor Ihnen. Schreiben Sie in dieses Skript nun Befehle, passiert jedoch zunächst einmal nichts – Sie müssen RStudio mitteilen, dass der von Ihnen geschriebene Code ausgeführt werden soll. Für diese Aktion stehen Ihnen drei Möglichkeiten zur Verfügung:
- Ausführen eines Codeteils: Hierzu markieren Sie den Part einer Zeile, den Sie ausführen möchten (Abbildung 5).
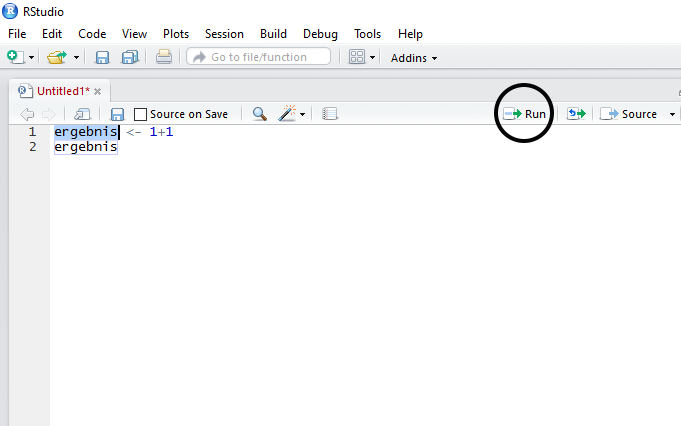
- Ausführen einer Codezeile: Hierzu setzen Sie den Cursor an eine beliebige Position in der Zeile (Abbildung 6).
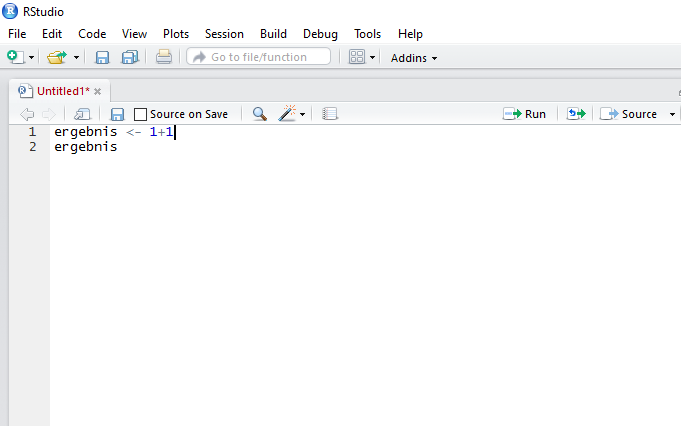
- Ausführen mehrerer Codezeilen: Hierzu markieren Sie alle Zeilen mit der Maus, die ausgeführt werden sollen (Abbildung 7).
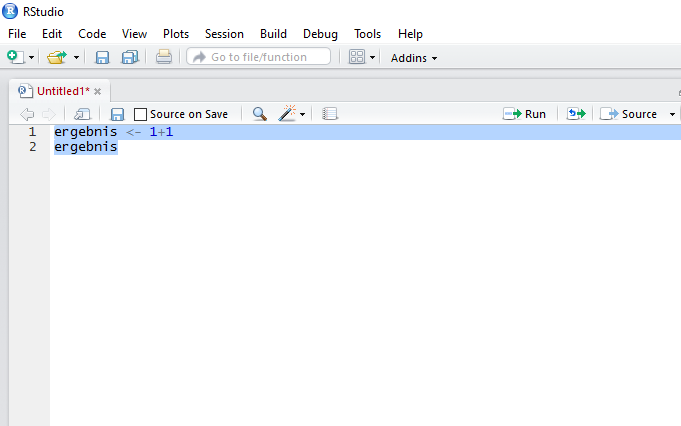
Auf jede dieser Varianten folgt derselbe Schritt: die
Tastenkombination [Strg] + [Enter]. Alternativ ist es auch
möglich, den Code über Run auszuführen (gekennzeichnet in Abbildung
5). Der Output erscheint daraufhin in der Konsole.
Ändern des Zielverzeichnisses
Im Regelfall importieren Sie Ihre Daten irgendwann in RStudio – und exportieren Ihre Ergebnisse im Laufe Ihrer Analyse. Damit dies nicht zu unangenehmen Überraschungen führt, weil Sie beispielsweise nicht wissen, wo Ihre kürzlich generierte Grafik liegt, müssen Sie RStudio sagen, in welchem Verzeichnis es standartmäßig nach einer Datei suchen soll. Dafür dient die Angabe eines Zielverzeichnisses. Das Zielverzeichnis definieren Sie auf eine der folgenden Weisen:
- die Tastenkombination
[Strg] + [Umschalt] + [H], - den Menüeintrag
Choose Directory, der überSession -> Set Working Directoryzu erreichen ist.
In beiden Fällen öffnet sich ein Dialogfenster, über das Sie Ihr
Zielverzeichnis festlegen und anschließend mit Choose
bestätigen können. Ein Blick auf die Konsole sagt Ihnen auch, welchen
Befehl RStudio im Hintergrund angewandt hat:
setwd("/home/users/sschneider")Das Zielverzeichnis soll hier also
/home/users/sschneider sein. Wenn Sie im weiteren Verlauf
wissen möchten, wie Ihr Zielverzeichnis lautet, wagen Sie einfach einen
Blick in das Panel Console: Dort ist das Zielverzeichnis in grauer
Schrift direkt neben dem Text Console im Titel hinterlegt.
Eine Alternative für Fortgeschrittene bietet folgender Befehl, der in
die Konsole einzugeben ist:
getwd()
## [1] "/home/users/sschneider"Auf das gewählte Zielverzeichnis bezieht sich RStudio nun in zwei Fällen, sofern kein absoluter Pfad angegeben wurde: Wenn eine Datei geladen oder gespeichert werden soll. Ein wichtiger Hinweis: Das Zielverzeichnis muss bei jedem Neustart von RStudio definiert werden. Angaben aus vorherigen Sitzungen werden nicht übernommen.
Die interne Hilfefunktion
In Kürze werden Sie bemerken, dass alle grundlegenden arithmetischen Funktionen bereits im Basispaket von RStudio vorhanden sind und Sie diese nur noch auszuführen brauchen. Dennoch kann es passieren, dass Sie nicht wissen, welche Argumente eine Funktion verlangt. Entsprechende Unterstützung bietet RStudio über die internen Hilfeseiten, die Sie über eine der folgenden Möglichkeiten aufrufen:
- den Befehl
help(funktionsname), - ein vorangestelltes Fragezeichen vor einer Funktion
(
?funktionsname), - die Suchfunktion des Reiters Help im Panel Working Directory, in
welche Sie den Funktionsnamen eintragen und mit
[Enter]bestätigen (beispielhaft in Abbildung 8).
Konkret bedeutet dies: Wenn Sie Informationen zu der Funktion
mean suchen, die den Durchschnitt mehrerer Zahlen
berechnet, nutzen Sie entweder die Suchfunktion oder führen Sie einen
der zwei Befehle aus:
help(mean)
?meanDie Hilfeseite öffnet sich anschließend im Reiter Help des Panels Working Directory. In den meisten Fällen finden Sie dort eine Beschreibung der Funktion (Description), wie sie aufgerufen wird (Usage), welche Argumente zugelassen sind (Arguments) und welcher Datentyp- beziehungsweise welche Datenstruktur zurückgegeben wird (Value). Weitere Informationen (Note) sind im Regelfall ebenso angeführt wie Referenzen zu entsprechender Literatur (References) und ein Block mit Beispielcode (Examples), den Sie über die Konsole ausführen können.
Anmerkungen
- Vielleicht erinnern Sie sich in Windows an den
Affengriff: Die Tastenkombination
[Strg] + [Alt] + [Entf]löst in neueren Versionen ein Menü mit Befehlen aus, um den Computer herunterzufahren oder den Taskmanager zu starten.↩︎ - Die deutschen Tastaturbelegungen finden Sie in der Wikipedia. Wenn Sie mit der Maus über die dort angezeigte Tastatur fahren, wird die Bezeichnung der jeweiligen Taste angezeigt.↩︎
Grundlagen in R
R ist eine objektorientierte, funktionale Programmiersprache. Oder um es in den Worten von John Chambers, Entwickler der Software S und ihres Nachfolgers R, zu sagen: „Alles, was existiert, ist ein Objekt. Alles, was passiert, ist ein Funktionsaufruf.“ 1 Ob Sie also mit einer Zahl arbeiten oder mit einer Zeichenkette, ist im übergeordneten Sinne irrelevant – eine Zahl ist ebenso ein Objekt wie eine Zeichenkette. Darauf aufbauend ist eine Feinabstufung möglich: Bei einem Objekt kann es sich um eine Funktion handeln – oder um Daten. Daten wiederum erlauben eine Untergliederung in Datentypen und Datenstrukturen, die beide jeweils weitere Verästelungen nach sich ziehen (Abbildung 1).
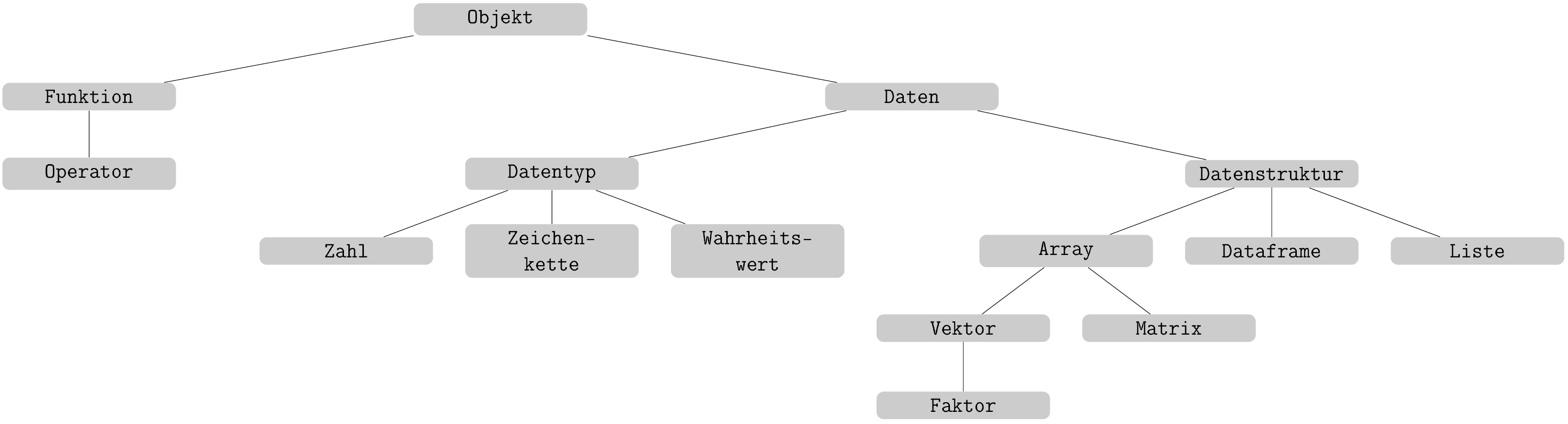
Objekte und Zuweisungen
Ein Objekt auf Datenebene ist demzufolge nichts anderes als ein
Auffangbehälter, der weitere Objekte unterschiedlichsten Typs und
unterschiedlichster Struktur beinhalten kann. 2 Für
den Anfang stellen Sie sich jedoch erst einmal ein Objekt vor, das aus
einem einzigen Wert (beispielsweise der Zahl 5) besteht.
Möchten Sie dieses Objekt später für weitere Berechnungen verwenden,
ohne permanent 5 eingeben zu müssen, können Sie ihm einen
Namen zuweisen:
x <- 5
x
## [1] 5
Umgangssprachlich ausgedrückt teilen Sie R mit: Nehme den Namen
x und übergebe ihm (<-) die Zahl
5. Das Objekt namens x enthält nun den Wert
5. Wie Sie durch Eingabe des vorhergehenden Codes
vielleicht schon bemerkt haben, gibt Ihnen R den Inhalt eines Objekts
nicht automatisch zurück – Sie müssen den Namen des Objekts in die
Konsole eingeben und ausführen (hier x in Zeile 2). Im
Prinzip speichern Sie ein Objekt durch Zuweisung eines Namens also immer
nach folgendem Schema ab: name <- objekt.
Namen müssen folgenden Vorschriften genügen:
- Sie dürfen keine Leerzeichen enthalten.
- Sie dürfen nicht mit einer Zahl beginnen (
1xist nicht erlaubt). - Auf Namen, die mit einem Punkt beginnen, darf keine Zahl folgen
(
.1xführt zu einer Fehlermeldung,.x1verursacht jedoch keine Probleme). - Sie dürfen nicht von R vorbelegt sein. 3
Bis auf diese Ausnahmen sind alle Buchstaben 4,
alle Zahlen und die Zeichen _ (Unterstrich) und
. (Punkt) erlaubt. Eine weitere Anmerkung: R unterscheidet
grundsätzlich zwischen Groß- und Kleinschreibung. Ein Objekt namens
A1 ist daher nicht identisch mit einem Objekt namens
a1. In jedem Fall ist es jedoch empfehlenswert, sich für
eine Konvention bei der Namenszuweisung zu entscheiden und diese im
gesamten Skript einzuhalten. Sie können beispielsweise eine dieser
Regeln verwenden:
- Namen bestehen nur aus Kleinbuchstaben. Bei mehreren Wörtern wird
kein Trennzeichen verwendet (
digitalhumanities). - Namen bestehen nur aus Kleinbuchstaben. Als Trennzeichen wird ein
Punkt (
digital.humanities) oder Unterstrich (digital_humanities) verwendet. - Namen mit einem Wort bestehen aus Kleinbuchstaben. Bei Namen mit
mehreren Wörtern wird das erste Wort klein geschrieben, die restlichen
Wörter leiten mit einem Großbuchstaben ein
(
digitalHumanities). - Namen bestehen aus Klein- und Großbuchstaben. Ein Wort wird durch
einen Großbuchstaben eingeleitet (
DigitalHumanities).
Datentypen
Ein Objekt lässt sich auf elementarster Ebene näher spezifizieren –
über seinen Datentyp. Die drei wichtigsten Datentypen in R lauten:
numeric, character und logical.
Handelt es sich bei Ihrem Objekt um eine Zahl, ordnen Sie den
entsprechenden Datentyp also vermutlich bereits intuitiv richtig zu:
numeric. Auch die weiteren zwei Typen sind annähernd
selbsterklärend: character ist der Container für
Zeichen (beispielsweise Buchstaben), logical der
Container für Wahrheitswerte (TRUE,
FALSE) 5.
Eine komprimierte Fassung finden Sie in nachfolgender Tabelle:
| Beschreibung | Beispiel | Datentyp |
|---|---|---|
| Ganze und reelle Zahlen | 3.17 |
numeric |
| Zeichen und Zeichenketten | "Bild" |
character |
| Wahrheitswerte | TRUE, FALSE |
logical |
Zwei Beispiele dürften Sie zurecht irritieren: Zum einen wird ein
Punkt als Dezimaltrennzeichen verwendet, zum anderen ist das Wort
Bild von Anführungszeichen umschlossen
("..."). Diese Fälle betrachten wir nun genauer. Zunächst
geben Sie die Zahl 3,17 in üblicher Notation mit Komma als
Dezimaltrennzeichen in die Konsole ein und führen die Zeile aus:
3,17
## Error: unexpected ',' in "3,Sie merken: RStudio gibt statt eines ordnungsgemäßen Outputs eine (durchaus kryptische) Fehlermeldung (Error) zurück. Um diese zu verstehen, müssen Sie sich bewusst machen, dass die Verwendung des Kommas in R vorbelegt ist: Das Zeichen kann Objekte eines Vektors ebenso abtrennen wie Argumente einer Funktion (mehr dazu in den nachfolgenden Abschnitten Datenstrukturen und Funktionen). Reelle Zahlen (Zahlen mit Nachkommastellen) benötigen daher in R einen Punkt statt eines Kommas als Dezimaltrennzeichen.
Sehen Sie sich nun das zweite Beispiele an, indem Sie
Bild in die Konsole eingeben und die Zeile ausführen:
Bild
## Error in eval(expr, envir, enclos): Objekt 'Bild' nicht gefundenAuch hier zeigt uns RStudio einen Error an, dieses Mal
jedoch mit einer konkreteren weiterführenden Information:
Objekt 'Bild' nicht gefunden. Die Erklärung ist simpel:
Ohne Anführungszeichen erwartet R beim Ausführen der Zeile ein Objekt
namens Bild, das im Workspace bereits existiert – von Ihnen
also, wie im Abschnitt Objekte und
Zuweisungen beschrieben, erzeugt wurde.
Achten Sie daher immer darauf, einen Wert mit Anführungszeichen
("Bild" oder 'Bild') zu umschließen, wenn es
sich bei ihm um Buchstaben oder eine Zeichenkette handelt und R diese
als solche erkennen soll.
Den Datentyp eines Objekts können Sie infolge über zwei Funktionen
bestimmen: mode und is.datentyp (also
is.numeric, is.character oder
is.logical). Der Unterschied: mode gibt den
genauen Datentyp zurück – is.datentyp prüft lediglich, ob
es sich um den von Ihnen spezifizierten Datentyp handelt und
spuckt einen Wahrheitswert aus. Sehen Sie sich dazu
beispielhaft folgenden Code an:
x <- 5
mode(x)
## [1] "numeric"
is.character(x)
## [1] FALSELaut mode ist das Objekt x vom Typ
numeric. Eine Prüfung, ob es sich bei x um ein
Objekt des Typs character handelt, resultiert folgerichtig
in der Rückgabe FALSE. Aber Achtung:
x <- "5"
mode(x)
## [1] "character"
is.character(x)
## [1] TRUESie weisen x im vorhergehenden Code durch die
umschließenden Anführungszeichen nicht mehr die Zahl sondern das Zeichen
5 zu – arithmetische Operationen wie im Abschnitt Operatoren können
mit diesem Objekt nicht mehr durchgeführt werden. Eine nachträgliche
Änderung des Datentyps ist über die Funktion as.datentyp
möglich:
x <- "5"
mode(x)
## [1] "character"
x <- as.numeric(x)
mode(x)
## [1] "numeric"Das Objekt x enthält zunächst wie bislang das Zeichen
5, dessen Datentyp Sie über mode testen. Im
Anschluss entscheiden Sie sich mit dem Aufruf
as.numeric(x), den Typ in numeric umzuwandeln
und das Ergebnis wieder in einem Objekt namens x abzulegen.
Die erneute Eingabe von mode sagt Ihnen daraufhin: Der
Datentyp des Objekts x ist numeric.
Die Umwandlung einer Zahl oder eines Zeichens in einen Wahrheitswert funktioniert hingegen nicht ohne Weiteres:
x <- "5"
x <- as.logical(x)
x
## [1] NADas Objekt x enthält nach der Änderung des Datentyps die
Angabe NA – statt eine Umwandlung des Zeichens
5 in einen Wahrheitswert vorzunehmen, hinterlegt R im
Objekt x die Konstante für einen fehlenden Wert,
NA (Not Available). NA kann in jeden
beliebigen Datentyp umgewandelt werden, ohne seine Form zu
verlieren:
x <- NA
as.numeric(x)
## [1] NA
as.character(x)
## [1] NA
as.logical(x)
## [1] NADatenstrukturen
Ein Level über den Datentypen liegen die Datenstrukturen: ein- bis
mehrdimensionale Beschreibungen über die (der Name verrät es bereits)
Struktur der Daten. Sie nehmen also Ihre bisherigen einzelnen Objekte
und bündeln sie – in Vektoren (vector), Matrizen
(matrix), Dataframes (data.frame) oder Listen
(list). Jede Struktur besitzt die grundlegende Eigenschaft
length. In mindestens zweidimensionalen Strukturen kommen
weitere Eigenschaften hinzu, die über die Funktion
attributes ermittelt werden können. Die Unterschiede und
Merkmale der Strukturen seien im Folgenden erläutert.
Vektoren
Die einfachste Datenstruktur in R, den Vektor, kennen Sie bereits, ohne es zu wissen. Denn alle Objekte, die nur einen Wert beinhalten, sind Vektoren der Länge 1. Stellen Sie sich einen Vektor jedoch nicht im schulmathematischen oder abstrakten Sinne vor, sondern beispielsweise als Stapel von Büchern, deren Titel Sie alphabetisch notieren. Die entstandene Liste würde bei Eingabe in R schließlich einen Vektor ergeben. Betrachten Sie zunächst allerdings noch einmal folgenden Output:
x <- "Bild"
x
## [1] "Bild"Vor der Zeichenkette "Bild" steht die Zahl
1 in eckigen Klammern: Es handelt sich um die Position des
Wertes "Bild" im Objekt x. Und dieses Objekt
x ist hier ein Vektor. Eine Information über die Länge
eines Objekts (also die Anzahl der in einem Objekt enthaltenen Elemente)
erhalten Sie mit der Funktion length:
length(x)
## [1] 1Nun gehen wir einen Schritt weiter und legen einen Vektor an, der mehr als ein Element enthält:
x <- c(2,3,5,7)
x
## [1] 2 3 5 7
length(x)
## [1] 4Betrachten Sie die erste Zeile und insbesondere den Befehl
c(2,3,5,7): Die Funktion c (kurz für
combine oder concatenate) legt mehrere durch Kommata
getrennte Werte (hier die Zahlen 2,3,5,7) als Vektor ab
(hier in das Objekt x). Anschließend geben Sie den Inhalt
von x aus. Es fällt auf: Die Markierung [1]
wird für alle folgenden Werte nicht weitergeführt – sie steht
ausschließlich am Anfang jeder Zeile des Outputs. Die Länge des
Vektors wird daraufhin geprüft: x enthält (wenig
überraschend) vier Elemente.
Wenn Sie den Wert eines bestimmten Elements in einem Vektor extrahieren möchten, übergeben Sie die Position des besagten Elements nach dem Namen des Vektors in eckigen Klammern (hier das Element mit Index 3):
x[3]
## [1] 5Mehrere Elemente können ebenso angesprochen werden. Handelt es sich um in einem Vektor aufeinander folgende Werte, verknüpfen Sie Start- und Endindex mit einem Doppelpunkt (hier die Elemente mit Index 2 bis 4):
x[2:4]
## [1] 3 5 7Handelt es sich hingegen um Elemente, die in einem Vektor nicht aufeinander folgen, müssen Sie dem Vektor einen weiteren Vektor mit den entsprechenden Indizes übergeben (hier die Elemente mit Index 2 und 4):
x[c(2,4)]
## [1] 3 7Warum dies nur scheinbar umständlich möglich ist (und nicht
beispielsweise über den Befehl x[2,4] abgedeckt wird),
bemerken Sie in Kürze, wenn Sie zweidimensionale Strukturen
kennenlernen. Die Selektion einzelner Elemente mag Ihnen übrigens
trivial und in diesem Stadium unnütz erscheinen – je mehr Elemente ein
Vektor (oder eine Datenstruktur allgemein) allerdings besitzt, umso
schwerer wird es Ihnen fallen, ein für Sie relevantes Element im
betreffenden Vektor zu finden.
Eine weitere und überaus relevante Eigenschaft dieser Datenstruktur: Ein Vektor kann nur Elemente desselben Datentyps aufnehmen. Probieren Sie folgendes:
x <- c("Bild", 3)
x
## [1] "Bild" "3"Da R die Zeichenkette "Bild" schlecht zu einer Zahl
umfunktionieren kann, wird der Datentyp der Zahl 3 geändert
– in das Zeichen 3. Den Datentyp aller in einem Vektor
enthaltenen Elemente erhalten Sie wie bislang über die Funktion
mode:
mode(x)
## [1] "character"Ein Vektor kann ferner vorne und hinten durch beliebig viele weitere Elemente ergänzt werden:
x <- c(2,3,5,7)
x
## [1] 2 3 5 7
x <- c("Bild", x, 3)
x
## [1] "Bild" "2" "3" "5" "7" "3"Kurzum können wir daher sagen: Ein Vektor ist eine geordnete Sammlung von Objekten mit gleichem Datentyp.
An dieser Stelle ist auch die Einführung eines weiteren Datentyps
sinnvoll: der des Faktors. Dieser Datentyp erleichtert die Arbeit mit
qualitativen Merkmalen, die verschiedene Merkmalsausprägungen besitzen.
Stellen Sie sich folgendes, stark vereinfachtes Beispiel vor: Ein Film
kann exakt einem von drei Genres zugeordnet werden – Drama, Komödie,
Thriller. In einem Vektor des Datentyps character, der
Informationen über acht Filme enthält, sieht dies möglicherweise so
aus:
x <- c("Komödie", "Komödie", "Drama", "Thriller", "Drama", "Komödie", "Drama", "Drama")
x
## [1] "Komödie" "Komödie" "Drama" "Thriller" "Drama" "Komödie" "Drama" "Drama"Für weitere Analysen ist jene Ausgangssituation jedoch unpraktisch: R
wird aus einem Vektor dieser Art nur beschwerlich eine simple Tabelle
mit den absoluten Häufigkeiten pro Genre erstellen können; Ihre
Merkmalsausprägungen werden nicht als solche erkannt – sie sind reiner
Text ohne zusätzliche Eigenschaften. Überführen Sie den Datentyp des
Vektors daher mit dem Befehl factor zu einem Faktor:
x <- factor(x)
x
## [1] Komödie Komödie Drama Thriller Drama Komödie Drama Drama
## Levels: Drama Komödie ThrillerDie erste Zeile des Outputs ist Ihnen bereits bekannt: Sie
gibt die Inhalte des Vektors wieder. In der zweiten Zeile erhalten Sie
hingegen eine neue Information: Ihr Vektor besitzt die drei Ausprägungen
(Levels) Drama, Komödie und
Thriller. Nützlich wird hier auch die Funktion
str, welche die Struktur Ihres Vektors feinstufiger
abbildet:
str(x)
## Factor w/ 3 levels "Drama","Komödie",..: 2 2 1 3 1 2 1 1Dass Sie einen Vektor des Datentyps factor mit drei
Ausprägungen erzeugt hatten, wussten Sie bereits. Der Output
zeigt Ihnen allerdings auch, wie R mit diesen Ausprägungen umgeht: Sie
werden zu Zahlen umkodiert (1,2,3), die einen Namen
besitzen ("Drama","Komödie","Thriller"). Jene sogenannten
kategorialen Variablen ermöglichen es Ihnen, R die von Ihnen vorgesehene
Gruppierung zu übergeben. Insbesondere bei numerisch hinterlegten,
qualitativen Ausprägungen besteht ansonsten die Gefahr, sie mit einer
quantitativen Variable zu verwechseln. 6
Machen Sie sich den Unterschied bewusst, indem Sie mit der Funktion
mean das arithmetische Mittel eines Vektors vom Typ
numeric und eines Vektors vom Typ factor
berechnen:
x <- c(1,1,4,5,4)
mean(x)
## [1] 3
y <- factor(x)
mean(y)
## Warning in mean.default(y): argument is not numeric or logical: returning NA
## [1] NAFür Vektoren vom Typ factor ist eine Berechnung des
arithmetischen Mittels (ebenso wie viele weitere Berechnungen) nicht
möglich – Abstände zwischen Faktoren sind nicht im üblichen numerischen
Sinne interpretierbar. 7 Ob es
sich um einen Faktor handelt,
können Sie wie bislang prüfen:
x <- c(1,1,4,5,4)
x <- factor(x)
is.factor(x)
## [1] TRUEDie Ausprägungen können Sie ferner auch separat über den Befehl
levels einsehen:
levels(x)
## [1] "1" "4" "5"Und in diesem Zuge auch verändern:
levels(x) <- c(3,4,5)
x
## [1] 3 3 4 5 4
## Levels: 3 4 5Entfernen Sie nun im Vektor x die Ausprägung
5 an Stelle 4:
x <- x[-4]
x
## [1] 3 3 4 4
## Levels: 3 4 5Die Information über die Ausprägung 5 bleibt im Vektor
also enthalten, obwohl in ihm keine Werte dieser Ausprägung mehr
vorhanden sind. Möchten Sie die Ausprägung ebenso entfernen, übergeben
Sie dem Vektor das Argument drop=TRUE:
x <- x[drop=TRUE]
x
## [1] 3 3 4 4
## Levels: 3 4Matrizen
Gehen wir einen Schritt weiter: Statt eindimensionalen Vektoren widmen wir uns jetzt zweidimensionalen Matrizen. Zweidimensional heißt im anschaulichen Sinne nichts anderes als: Es wird eine Tabelle mit Zeilen (waagerecht) und Spalten (senkrecht) aufgespannt. In Code ausgedrückt bedeutet dies:
x <- matrix(data=5:10, nrow=3, ncol=2)
x
## [,1] [,2]
## [1,] 5 8
## [2,] 6 9
## [3,] 7 10Mit der Funktion matrix teilen Sie R mit, eine Matrix zu
erstellen, die in diesem Fall mit den Werten 5 bis 10
(data=5:10) über drei Zeilen (nrow=3) und zwei
Spalten (ncol=2) bestückt werden soll. Die Argumente der
Funktion werden durch Kommata abgetrennt. Die Reihenfolge der Argumente
spielt im Grunde keine Rolle, wenn Sie die entspechenden Argumente nicht
abkürzen (also matrix(5:10, 3, 2) statt
matrix(data=5:10, nrow=3, ncol=2) schreiben).
Der Output macht Ihnen auch deutlich: Die Daten, bestehend
aus den Zahlen 5 bis 10, füllen die Matrix spaltenweise auf – zunächst
wird die erste Spalte vervollständigt (im Output markiert durch
[,1]), dann die zweite Spalte ([,2]). Diese
Voreinstellung ändern Sie, indem Sie der Funktion matrix
das Argument byrow=TRUE übergeben:
x <- matrix(data=5:10, nrow=3, ncol=2, byrow=TRUE)
x
## [,1] [,2]
## [1,] 5 6
## [2,] 7 8
## [3,] 9 10Eine Matrix hat zudem immer auch eine Dimension, welche über die
Funktion dim ausgegeben werden kann:
dim(x)
## [1] 3 2Die Dimension der Matrix x besteht aus der Anzahl der
Zeilen an Position 1 (3) und der Anzahl der Spalten an
Position 2 (2). Diese Angaben erhalten Sie zudem mit den
Befehlen nrow und ncol separat:
nrow(x)
## [1] 3
ncol(x)
## [1] 2Die Länge derselben Matrix bildet sich folglich aus der Multiplikation der Anzahl der Zeilen mit der Anzahl der Spalten:
length(x)
## [1] 6Was passiert nun aber, wenn sich die Anzahl der Elemente, die in eine Matrix eingefügt werden sollen, von der Dimension der aufzuspannenden Matrix unterscheidet? Sehen Sie selbst:
matrix(data=5:11, nrow=3, ncol=2)
## [,1] [,2]
## [1,] 5 8
## [2,] 6 9
## [3,] 7 10
## Warning in matrix(5:11, nrow = 3, ncol = 2): data length [7] is not a sub-multiple or multiple of the number of rows [3]Im vorhergehenden Fall versuchen Sie, einen Vektor der Länge 7 (die
Zahlen 5 bis 11) in einer Matrix der Länge 6 unterzubringen: R gibt eine
Warnung zurück (Warning) und ignoriert das Element an Position
7 des Vektors (die Zahl 11), da 7 kein Vielfaches von 3
(der Anzahl der Zeilen) ist – es werden wie bislang nur die Werte 5 bis
10 an die Matrix übergeben.
Für den Fall, dass die Anzahl der einzufügenden Elemente die Länge der Matrix unterschreitet, ergibt sich folgendes Bild:
matrix(data=5:9, nrow=3, ncol=2)
## [,1] [,2]
## [1,] 5 8
## [2,] 6 9
## [3,] 7 5
## Warning in matrix(5:9, nrow = 3, ncol = 2): data length [5] is not a sub-multiple or multiple of the number of rows [3]Das erste Element des Vektors (die Zahl 5) wird in
letzterem Beispiel recycelt – es tritt in Ermangelung eines
weiteren Elements an Position 10 des Vektors. Damit ist das Resultat
identisch zu folgendem Befehl:
matrix(data=c(5:9,5), nrow=3, ncol=2)
## [,1] [,2]
## [1,] 5 8
## [2,] 6 9
## [3,] 7 5Kommen wir zu der Selektion eines oder mehrerer Elemente in einer
Matrix. Die Struktur lässt sich im zweidimensionalen Fall mit folgendem
Schema beschreiben: matrix[zeile,spalte]. Das Element in
der zweiten Zeile der ersten Spalte erhalten Sie daher mit dem
Befehl:
x[2,1]
## [1] 7Alle Regelungen für die Indexierung eines Elements in einem Vektor gelten analog auf Zeilen- und Spaltenebene in einer Matrix. Möchten Sie die Werte einer bestimmten Spalte extrahieren, müssen Sie keine Angabe über die benötigten Zeilen treffen:
x[,1]
## [1] 5 7 9Der Befehl gibt Ihnen die erste Spalte der Matrix x
zurück. Wenn Sie Interesse an den Werten der zweiten Zeile haben, gehen
Sie wie folgt vor:
x[2,]
## [1] 7 8Um den Umgang zu erleichtern, ist es zudem möglich, den Zeilen und Spalten Namen zuzuweisen. Entweder spezifizieren Sie diese zusätzlichen Informationen gleich bei Anlage der Matrix:
x <- matrix(data=5:10, nrow=3, ncol=2, dimnames=list(c("Zeile1", "Zeile2", "Zeile3"), c("Spalte1", "Spalte2")))
x
## Spalte1 Spalte2
## Zeile1 5 8
## Zeile2 6 9
## Zeile3 7 10Oder im Nachhinein mit der Funktion dimnames:
x <- matrix(data=5:10, nrow=3, ncol=2)
dimnames(x) <- list(c("Zeile1", "Zeile2", "Zeile3"), c("Spalte1", "Spalte2"))
x
## Spalte1 Spalte2
## Zeile1 5 8
## Zeile2 6 9
## Zeile3 7 10In beiden Fällen übergeben Sie dimnames eine Liste, die
aus einem Vektor mit den Zeilennamen und einem Vektor mit den
Spaltennamen besteht. Weiteres zum Thema Listen finden Sie am Ende
dieses Abschnitts. Über die vergebenen Namen können Sie ebenso auf die
Zeilen und Spalten zugreifen:
x["Zeile3",]
## Spalte1 Spalte2
## 7 10
x[,"Spalte2"]
## Zeile1 Zeile2 Zeile3
## 8 9 10Natürlich können auf diese Weise auch mehrere Zeilen oder Spalten angesprochen werden, indem Sie die gewünschten Zeilen- oder Spaltennamen an einen Vektor übergeben:
x[c("Zeile2", "Zeile3"),]
## Spalte1 Spalte2
## Zeile2 6 9
## Zeile3 7 10Ebenso ist es über die Befehle rownames beziehungsweise
colnames möglich, nur die Zeilen- beziehungsweise
Spaltennamen abzufragen:
rownames(x)
## [1] "Zeile1" "Zeile2" "Zeile3"
colnames(x)
## [1] "Spalte1" "Spalte2"Möchten Sie nun den Namen der zweiten Zeile ändern, übergeben Sie
einfach den entsprechenden Index an den Vektor der Zeilennamen
(rownames(x)) und weisen ihm den neuen Wert zu
("ZeileZwei"):
rownames(x)[2] <- "ZeileZwei"
rownames(x)
## [1] "Zeile1" "ZeileZwei" "Zeile3"Wenn Sie eine weitere Spalte benötigen, die bei Erstellen der Matrix
noch nicht berücksichtigt wurde, hilft Ihnen die Funktion
cbind (column bind), an die Sie die bisherige
Matrix und einen Vektor mit den Werten der neuen Spalte übergeben:
y <- cbind(x, c(15:17))
y
## Spalte1 Spalte2
## Zeile1 5 8 15
## ZeileZwei 6 9 16
## Zeile3 7 10 17Mit dem Befehl rbind (row bind) fügen Sie
analog eine neue Zeile hinzu:
z <- rbind(y, c(11:13))
z
## Spalte1 Spalte2
## Zeile1 5 8 15
## ZeileZwei 6 9 16
## Zeile3 7 10 17
## 11 12 13In beiden Fällen fällt auf: Über rbind beziehungsweise
cbind wird bei Anlage einer neuen Zeile beziehungsweise
Spalte nicht automatisch ein entsprechender Zeilenbeziehungsweise
Spaltenname generiert. Diesen müssen Sie manuell übergeben:
rownames(z)[4] <- "Zeile4"
rownames(z)
## [1] "Zeile1" "ZeileZwei" "Zeile3" "Zeile4"
colnames(z)[3] <- "Spalte3"
colnames(z)
## [1] "Spalte1" "Spalte2" "Spalte3"
z
## Spalte1 Spalte2 Spalte3
## Zeile1 5 8 15
## ZeileZwei 6 9 16
## Zeile3 7 10 17
## Zeile4 11 12 13Eine Matrix ist somit vergleichbar mit einem zweidimensionalen Vektor und nimmt wie dieser nur Objekte gleichen Datentyps auf.
Dataframes
Bei spaltenweise unterschiedlichen Datentypen empfiehlt sich im zweidimensionalen Fall die Verwendung eines Dataframes – eine Spalte kann dort beispielsweise Zahlen aufnehmen, eine andere Zeichenketten. Wie bisher gilt: Die einzelnen Spalten eines Dataframes weisen dieselbe Länge auf, fehlende Werte müssen explizit gekennzeichnet werden. Generieren wir nun einen Dataframe aus der Sequenz der Zahlen 5 bis 10 mit denselben Argumenten, die wir bei der ursprünglichen Beispielmatrix verwendet haben:
x <- data.frame(data=5:10, nrow=3, ncol=2)
x
## data nrow ncol
## 1 5 3 2
## 2 6 3 2
## 3 7 3 2
## 4 8 3 2
## 5 9 3 2
## 6 10 3 2Dataframes funktionieren offensichtlich anders als Matrizen: Jedes
zusätzliche Argument der Funktion data.frame resultiert in
einer neuen Spalte. Der Spaltenname wird aus der Angabe vor dem
= extrahiert (beispielsweise data), die Daten
wie bislang aus der Angabe nach dem = (beispielsweise
5:10). Wenn Sie also einen Dataframe ähnlich der
vorhergehenden Matrix aufspannen möchten, übergeben Sie die Werte
spaltenweise:
x <- data.frame(Spalte1=c(5:7), Spalte2=c(8:10))
x
## Spalte1 Spalte2
## 1 5 8
## 2 6 9
## 3 7 10Die Dimension eines Dataframes ist, bei gleicher Befüllung, identisch zu der Dimension einer Matrix:
dim(x)
## [1] 3 2Passen Sie jedoch bei dem Befehl length auf:
length(x)
## [1] 2Die Länge eines Dataframes ergibt sich nicht wie im Falle einer
Matrix aus der Multiplikation der Anzahl der Zeilen und Spalten, sondern
– möglicherweise ahnen Sie es bereits – nur aus der Anzahl der Spalten.
Die Befehle nrow und ncol können jedoch wie
bislang angewandt werden:
nrow(x)
## [1] 3
ncol(x)
## [1] 2Ebenso ohne Unterschiede verhält sich das Prinzip des
Recycling sowie die Befehle rownames und
colnames (die Spaltennamen lesen wir ausnahmsweise nur aus
und verändern sie nicht):
rownames(x) # Zeilennamen vorher
## [1] "1" "2" "3"
rownames(x) <- c("Zeile1", "Zeile2", "Zeile3")
rownames(x) # Zeilennamen nachher
## [1] "Zeile1" "Zeile2" "Zeile3"
colnames(x)
## [1] "Spalte1" "Spalte2"
x
## Spalte1 Spalte2
## Zeile1 5 8
## Zeile2 6 9
## Zeile3 7 10Zusätzlich zu der Ihnen bekannten Selektionsform für zweidimensionale
Datenstrukturen (über object[zeile,spalte]) bietet ein
Dataframe für Spalten auch die praktischere Selektion über den
Dollar-Operator an:
x$Spalte2
## [1] 8 9 10Dieselbe Methode funktioniert allerdings nicht auf Zeilenebene:
x$Zeile3 # Neue Variante
## NULL
x["Zeile3",] # Alte Variante
## Spalte1 Spalte2
## Zeile3 7 10Ein Element einer Spalte kann folglich wieder mit den entsprechenden Indizes extrahiert werden:
x$Spalte2[2]
## [1] 9Beachten Sie jedoch, dass dieser Operator nicht für Matrizen zur Verfügung steht:
y <- matrix(c(5:7, 8:10), nrow = 3, ncol = 2)
colnames(y) <- c("Spalte1", "Spalte2")
y
## Spalte1 Spalte2
## [1,] 5 8
## [2,] 6 9
## [3,] 7 10
y$Spalte1
## Error in y$Spalte1 : $ operator is invalid for atomic vectorsWarum in diesem Fall eine Fehlermeldung erscheint, teilt Ihnen die
interne Hilfefunktion bei Aufruf des Befehls ?"$" mit: „$
is only valid for recursive objects [...].“ Handelt es sich bei unserer
Matrix y um ein rekursives Objekt?
is.recursive(y)
## [1] FALSEWenn y kein rekursives Objekt ist und somit
keine listenähnliche Struktur aufweist, müsste es stattdessen ein
atomares Objekt sein:
is.atomic(y)
## [1] TRUEDa eine Matrix, wie Sie am Anfang dieses Abschnitts bemerkt haben, auf Vektoren aufsetzt, ist der Dollar-Operator für diese in R nicht implementiert. Ein Dataframe hingegen bildet eine Sonderform der demnächst erläuterten Datenstruktur der Liste aus – und kann demzufolge den Dollar-Operator nutzen.
Spannen Sie nun einen Dataframe auf, der in einer Spalte Elemente des
Typs character und in einer weiteren Spalte Elemente des
Typs numeric enthält:
artist <- data.frame(Name = c("Peter Paul Rubens", "Ludwig Mies van der Rohe", "Adolph Menzel"), Geburtsjahr = c(1619, 1950, 1851))
artist
## Name Geburtsjahr
## 1 Peter Paul Rubens 1619
## 2 Ludwig Mies van der Rohe 1950
## 3 Adolph Menzel 1851Fügen Sie jetzt einen weiteren Künstler hinzu:
artist[4,1] <- "William Poole"
## Warning in ‘[<-.factor‘(‘*tmp*‘, iseq, value = "William Poole"): invalid factor level, NA generatedMerkwürdig, oder? Prüfen Sie doch einmal den Datentyp der Spalte
Name:
mode(artist$Name)
## [1] "numeric"Der Befehl mode scheint uns nicht zu helfen – testen Sie
daher, um welche Klasse es sich bei der Spalte handelt:
class(artist$Name)
## [1] "factor"Da Faktoren in R keine atomaren Datentypen sind, sie sich also in
numerische Werte weiter zerlegen lassen, ist zur Erkennung eines Faktors
der Befehl class vonnöten. Warum aber verursacht die
eigentlich simple Aktion, einen weiteren Künstler mit Name und
Geburtsjahr hinzufügen zu wollen, solche Probleme? Die Antwort:
Standardmäßig legt R Spalten eines Dataframes mit dem Datentyp
factor an. Entweder Sie erstellen den Dataframe nochmals
mit dem zusätzlichen Argument stringsAsFactors = FALSE und
fügen im Anschluss den neuen Künstler hinzu:
artist <- data.frame(Name = c("Peter Paul Rubens", "Ludwig Mies van der Rohe", "Adolph Menzel"), Geburtsjahr = c(1577, 1886, 1815), stringsAsFactors = FALSE)
artist
## Name Geburtsjahr
## 1 Peter Paul Rubens 1577
## 2 Ludwig Mies van der Rohe 1886
## 3 Adolph Menzel 1815
artist[4,1] <- "William Poole"
artist[4,2] <- 1774
artist
## Name Geburtsjahr
## 1 Peter Paul Rubens 1577
## 2 Ludwig Mies van der Rohe 1886
## 3 Adolph Menzel 1815
## 4 William Poole 1774Oder Sie wandeln stattdessen die Spalte Name in den
Datentyp character um:
artist$Name <- as.character(artist$Name)
artist[4,1] <- "William Poole"
artist[4,2] <- 1774
artist
## Name Geburtsjahr
## 1 Peter Paul Rubens 1577
## 2 Ludwig Mies van der Rohe 1886
## 3 Adolph Menzel 1815
## 4 William Poole 1774Mithilfe des Dollar-Operators können Sie im Übrigen auch eine neue Spalte erstellen, sofern der übergebene Spaltenname noch nicht vergeben wurde:
artist$Geschlecht <- "männlich"
artist
## Name Geburtsjahr Geschlecht
## 1 Peter Paul Rubens 1577 männlich
## 2 Ludwig Mies van der Rohe 1886 männlich
## 3 Adolph Menzel 1815 männlich
## 4 William Poole 1774 männlichWidmen wir uns endlich fortgeschrittenen Analysemethoden – nämlich der Selektion von Teilmengen. Eine Möglichkeit ist die bereits ausgeführte Form der Indizierung:
artist[artist$Name == "Adolph Menzel",]
## Name Geburtsjahr Geschlecht
## 3 Adolph Menzel 1815 männlichÜber artist$Name == "Adolph Menzel" stellen Sie fest,
welche Zellen der Spalte Name im Dataframe artist exakt
(==) 8 den Inhalt
"Adolph Menzel" aufweisen:
artist$Name == "Adolph Menzel"
## [1] FALSE FALSE TRUE FALSEDas Prozedere ist daher folgendes: R nimmt zunächst den Künstlernamen
an Position 1 des Vektors (Peter Paul Rubens) und gleicht
ihn mit dem anschließend notierten Namen (Adolph Menzel)
ab. Liegt eine Übereinstimmung vor, gibt R den Wahrheitswert
TRUE zurück, liegt keine Übereinstimmung vor, den
Wahrheitswert FALSE. Solange nicht geprüfte Elemente im
Vektor artist$Name vorhanden sind, wird dieser Vorgang für
jedes verbleibende Element wiederholt.
Laut Output kommt der gesuchte Name nicht
(FALSE) in den Zellen 1, 2 und 4 vor. Zelle 3 jedoch
liefert einen Treffer durch die Rückgabe TRUE zurück. Die
genaue Position des mit Wahrheitswert TRUE hinterlegten
Wertes erhalten Sie ebenso über den Befehl which, der Ihre
bisherige Abfrage umschließt:
which(artist$Name == "Adolph Menzel")
## [1] 3Demzufolge ist Ihre Selektionsabfrage nichts anderes als:
artist[3,]
## Name Geburtsjahr Geschlecht
## 3 Adolph Menzel 1815 männlichWenn Sie nun jene Zeilen ausgeben möchten, welche die Künstler Adolph Menzel und Peter Paul Rubens enthalten, würden Sie annehmen, dass dies mit folgendem Befehl möglich ist:
artist[artist$Name == c("Peter Paul Rubens", "Adolph Menzel"),]
## Name Geburtsjahr Geschlecht
## 1 Peter Paul Rubens 1577 männlichDer Operator == wertet allerdings nur eine
Übereinstimmung mit dem ersten Element des Vektors
c("Peter Paul Rubens", "Adolph Menzel") aus. Daher müssen
Sie einen anderen Operator einsetzen:
artist[artist$Name %in% c("Peter Paul Rubens", "Adolph Menzel"),]
## Name Geburtsjahr Geschlecht
## 1 Peter Paul Rubens 1577 männlich
## 3 Adolph Menzel 1815 männlichJedes Element des Vektors artist$Name wird nun gegen die
Menge c("Peter Paul Rubens", "Adolph Menzel") auf
Übereinstimmung geprüft. Aber auch diese Methode hilft Ihnen nicht
weiter, sofern Sie an einzelnen Zellbestandteilen interessiert sind.
Dafür eignet sich jedoch die Funktion grepl:
artist[grepl("en", artist$Name),]
## Name Geburtsjahr Geschlecht
## 1 Peter Paul Rubens 1577 männlich
## 3 Adolph Menzel 1815 männlichDiese Funktion ist eine Allzweckwaffe, wenn es darum geht, Elemente
jeglicher Form auf vorhandene Zeichenketten oder Muster zu überprüfen –
es ist sogar möglich, sie mit regulären Ausdrücken zu bestücken
9.
Im vorliegenden Fall suchen Sie
nach der Zeichenfolge en im Vektor artist$Name
und erhalten zwei Ergebnisse: die Künstler Peter Paul Rubens und Adolph
Menzel.
Ein Dataframe ist somit vergleichbar mit einer Matrix. Im Gegensatz zu dieser nimmt er jedoch spaltenweise Objekte unterschiedlichen Datentyps auf und ist aus diesem Grund die für Datenanalysen am Häufigsten genutzte Datenstruktur. 10
Listen
Um aus unseren bislang maximal zweidimensionalen Strukturen
auszubrechen oder unterschiedliche Strukturen effektiv verknüpfen zu
können, bietet R eine weitere Struktur an: die Liste. Eine Liste ist im
Grunde nichts anderes als ein Container, in dem sich andere
Container befinden – beispielsweise Vektoren, Matrizen und
Dataframes. Sie wird über den Befehl list
initialisiert:
liste <- list()
liste
## list()In diesem Zuge kann sie ebenso befüllt werden:
liste <- list(c("Buch", "Zeitschrift"), c(1:4), matrix(11:14, 2))
liste
## [[1]]
## [1] "Buch" "Zeitschrift"
##
## [[2]]
## [1] 1 2 3 4
##
## [[3]]
## [,1] [,2]
## [1,] 11 13
## [2,] 12 14Im Output sehen Sie, dass die Elemente der soeben kreierten Liste jeweils über doppelte eckige Klammern indiziert werden. Folglich können Sie über diese auch die Inhalte der Elemente extrahieren:
liste[[2]]
## [1] 1 2 3 4Oder Sie benennen sie zunächst:
liste <- list(Element1 = c("Buch", "Zeitschrift"), Element2 = c(1:4), Element3 = matrix(11:14, 2))
liste
## $Element1
## [1] "Buch" "Zeitschrift"
##
## $Element2
## [1] 1 2 3 4
##
## $Element3
## [,1] [,2]
## [1,] 11 13
## [2,] 12 14Und nutzen infolge wie gehabt den Dollar-Operator:
liste$Element3
## [,1] [,2]
## [1,] 11 13
## [2,] 12 14Eine spezifische Spalte der Matrix Element3 selektieren
Sie nun durch eine Kombination der bislang erlernten Befehle:
liste$Element3[,2]
## [1] 13 14Fügen Sie der Liste doch auch noch einen Dataframe hinzu:
liste$Element4 <- data.frame(Kinostart = c("26.05.2016", "02.06.2016"), Titel = c("Der Nachtmahr", "The Nice Guys"))
liste
## $Element1
## [1] "Buch" "Zeitschrift"
##
## $Element2
## [1] 1 2 3 4
##
## $Element3
## [,1] [,2]
## [1,] 11 13
## [2,] 12 14
##
## $Element4
## Kinostart Titel
## 1 26.05.2016 Der Nachtmahr
## 2 02.06.2016 The Nice GuysMit diesem testen Sie jetzt, ob eine Selektion über zwei nacheinander deklarierte Dollar-Operatoren funktioniert:
liste$Element4$Titel
## [1] Der Nachtmahr The Nice Guys
## Levels: Der Nachtmahr The Nice GuysMehrere Elemente einer Liste können auf bekanntem Wege durch Übergabe eines Vektors mit den entsprechenden Namen der Elemente angewählt werden:
liste[c("Element1", "Element2")]
## $Element1
## [1] "Buch" "Zeitschrift"
##
## $Element2
## [1] 1 2 3 4Wie aber wählen Sie mehrere Elemente über deren Indizes aus?
liste[[1:2]]
## [1] "Zeitschrift"Diese Variante funktioniert offensichtlich nicht. Die Ihnen bekannte Möglichkeit über einfache eckige Klammern aber schon:
liste[1:2]
## $Element1
## [1] "Buch" "Zeitschrift"
##
## $Element2
## [1] 1 2 3 4Eine Liste ermöglicht es demnach, unterschiedliche Datenstrukturen und deren Elemente in einem Objekt zu verknüpfen und auf diese weiterhin zugreifen zu können.
Funktionen
Coming Soon.
Operatoren
Coming Soon.
Anmerkungen
- Chambers, John M. (2014): Object-Oriented Programming, Functional Programming and R. In: Statistical Science. 29.2, S. 167-180, URL: http://arxiv.org/pdf/1409.3531↩︎
- Die Rolle der Funktionen sei vorerst außen vor gelassen, eine ausführliche Erklärung dieser finden Sie im Abschnitt Funktionen.↩︎
- Alle reservierten Namen finden Sie unter: https://stat.ethz.ch/R-manual/R-devel/library/base/html/Reserved.html.↩︎
- Sie könnten beispielsweise ein Objekt namens
âanlegen. Es sei allerdings aus Gründen des guten Stils davon abgeraten, Sonderzeichen zu benutzen.↩︎ - Ein Wahrheitswert gibt an, ob eine Aussage wahr
oder falsch ist. Die Aussage „Albrecht Dürer war ein Maler“
wäre demnach wahr (
TRUE).↩︎ - Quantitativ heißt im Grunde nichts anderes als: mit Zahlen zu erfassen. Die Frage, wie Ihnen ein Kunstwerk auf einer Skala von 1 bis 10 gefällt, fordert daher eine Zahl zwischen 1 und 10 als Antwort (quantitativ). Wenn Sie jedoch stattdessen gefragt werden, wie Sie ein Kunstwerk beschreiben würden, geben Sie eine persönliche Stellungsnahme ab, die nicht ins Numerische übertragen werden kann (qualitativ).↩︎
- Es wäre vermutlich auch etwas seltsam, den Durchschnitt aus Äpfeln und Birnen zu berechnen.↩︎
- Mit einem einzigen Gleichheitszeichen weisen Sie einem Argument einer Funktion also einen Wert zu, mit einem doppelten Gleichheitszeichen prüfen Sie, ob zwei Variablen übereinstimmen.↩︎
- Für weitere Informationen über die Natur und den Zweck regulärer Ausdrücke gehen Sie auf das entsprechenden Punkt links im Menü.↩︎
- Gänzlich ohne Nachteile sind die Freiheiten eines Dataframes jedoch nicht: Wenn komplexere Funktionen auf (vor allem umfangreichere) Dataframes angewandt werden, benötigen diese im Vergleich zu einer Anwendung auf Matrizen deutlich mehr Zeit.↩︎
Reguläre Ausdrücke
Zeichenfolgen können beschrieben, gesucht und ersetzt werden. Dies
erfolgt durch den Einsatz regulärer Ausdrücke
(Regular Expressions, RegEx).
Verwendung finden RegEx häufig in Webanwendungen (z.B. Perl, PHP) oder
in Unix-Skripten:
"Reguläre Ausdrücke (oder kurz: Regexps von engl. regular expressions)
stellen in der Programmierung verallgemeinerte Suchmuster dar. Mithilfe
dieser Suchmuster können Sie beispielsweise Variableninhalte durchsuchen
und bestimmte Inhalte daraus herausziehen oder ersetzen. [...] Reguläre
Ausdrücke sind auch ein mächtiges Mittel, um große Datenbestände nach
komplexen Suchausdrücken zu durchforsten. Beispielsweise könnten alle
Begriffe gesammelt werden, die mit „A“ beginnen und auf „tion“ oder
„tung“ enden, was mit gewöhnlichen Stringfunktionen nur mühselig zu
bewerkstelligen wäre."1
Einfache Ausdrücke
Eckige Klammern bezeichnen in einer
Zeichenfolge eine Alternative, z.B. einen alternativen Buchstaben oder
eine alternative Zahl. Einige kurze Beispiele zur Illustration:
"Hans": Ha[un]s -> Das Suchergebnis umfasst sowohl das Wort "Haus"
als auch den Namen "Hans".
"1990": 199[0-9] -> Das Suchergebnis gibt alle Jahre aus, die zum
entsprechenden Jahrzehnt gehören.
Grundsätzlich gilt: Es können beliebig viele
RegEx aufeinander folgen.
Ist beispielsweise nur das Jahrhundert bekannt, können die Jahrzehnte
und Jahre wie folgt umschrieben werden:
19[0-9][0-9] -> Das Suchergebnis gibt alle Jahre von 1900 bis 1999
an.
Ausgabe einer dreistelligen Zahl, deren Ziffern beliebig sein
können, jedoch ohne 0:
[1-9]{3} -> Das Suchergebnis gibt alle Zahlen aus, die zwischen 111
und 999 liegen.
Eine kleine Auswahl an weiteren gängigen Zeichen in RegEx:
| Zeichen | Funktion | Beispieleingabe | Beispielausgabe | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ? | kein oder genau ein Vorkommen | Ha?mmer | Hmmer oder Hammer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| + | mindestens ein oder beliebig viele Vorkommen | Ham+er | Hamer, Hammer, Hammmer, etc. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| * | kein oder beliebig viele Vorkommen | Ham*er | Haer, Hamer, Hammer, Hammmer, etc. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| {2} | exakt zwei Vorkommen | Ham{2}er | Hammer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| {1,3} | mindestens ein, maximal drei Vorkommen | Ham{1,3}er | Hamer, Hammer, Hammmer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
- \ maskiert die Sonderfunktion von Zeichen wie +, *, etc., um nach diesen Zeichen selbst suchen zu können.
- ^ markiert den Anfang eines zu durchsuchenden Strings ("ab hier").
- $ markiert das Ende eines zu durchsuchenden Strings ("bis hier").
(Ein) Online-Tool zum Testen von regulären Ausdrücken
vor dem Implementieren:
regex101.com
Ausführliche Informationen:
"Die
regulären Ausdrücke" in DH-Lehre
Ein
gutes RegEx-Tutorial
Liste der Funktionen
Hier finden Sie viele in den Modulen vorgestellten Funktionen, alphabetisch sortiert nach dem Anfangsbuchstaben des jeweiligen Funktionsaufrufs. Zu jeder Funktion ist der grundlegende Aufruf mit Argumenten und den jeweils vorbelegten Werten, eine Beschreibung des Aufrufs und (sofern vorhanden) ein Link zur Hilfeseite des R Project angegeben, über den Sie anschauliche Beispiele sowie Erläuterungen erhalten. Weitere nützliche Funktionen werden (ohne Anspruch auf Vollständigkeit) sukzessive ergänzt.
| Aufruf | Beschreibung | Hilfe | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
as.character(object) |
Ändern des Datentyps eines Objekts zu character. Ein
Objekt, das
nicht umgewandelt werden kann, erhält die Angabe NA.
|
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
as.logical(object) |
Ändern des Datentyps eines Objekts zu logical. Ein
Objekt, das nicht
umgewandelt werden kann, erhält die Angabe NA. |
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
as.numeric(object) |
Ändern des Datentyps eines Objekts zu numeric. Ein
Objekt, das nicht
umgewandelt werden kann, erhält die Angabe NA. |
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
c(...) |
Verknüpfen (combine, concatenate) mehrerer durch Kommata
getrennter
Objekte zu einem Objekt (beispielsweise c(object1,
object2)). |
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
cbind(...) |
Hinzufügen einer oder mehrerer Spalten (in Vektorform) zu einer
Matrix (beispielsweise cbind(matrix1, vector1)). Wenn
die Anzahl der Elemente des Vektors die Anzahl der Elemente in einer
Spalte der Matrix unterschreitet, wird der letzte
Wert des Vektors recycelt. |
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
colnames(object) |
Ausgabe der Spaltennamen eines Objekts, bei dem es sich um eine Matrix oder um einen Dataframe handelt. | ? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
data.frame(...) |
Erstellen eines Dataframes. Über ... kann eine
beliebige Anzahl von
Spalten mit deren Namen und Werten nach dem Schema
name = werte übergeben werden. Ein Dataframe mit zwei
Spalten,
welche die Namen Spalte1 und Spalte2
tragen und mit den
Werten c(1:3) und c(4:6) befüllt werden
sollen, wird folgendermaßen
aufgespannt: data.frame(Spalte1 = c(1:3), Spalte2 =
c(4:6)).
|
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
dim(object) |
Ausgabe der Dimension eines Objekts. Im Falle einer zweidimensionalen Matrix steht zuerst die Anzahl der Zeilen, dann die Anzahl der Spalten. | ? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
dimnames(object) |
Ausgabe der Dimensionsnamen eines Objekts. Im Falle einer
zweidimensionalen Matrix stehen zuerst die Namen der Zeilen,
dann die Namen der Spalten. Ebenso können die Namen der Zeilen und
Spalten über dimnames in einer Liste von Vektoren
übergeben werden (beispielsweise dimnames <-
list(c("Zeile1", "Zeile2" , "Zeile3" ), c("Spalte1", "Spalte2"
))). |
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
getwd() |
Ausgabe des aktuellen Arbeits- beziehungsweise Zielverzeichnisses. | ? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
help(function) |
Ausgabe der internen Hilfeseite einer Funktion (beispielsweise von
der Funktion zur Berechnung des Mittelwerts mit
help(mean)
). Alternativ kann die interne Hilfe über ?function
aktiviert werden.
|
- | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
is.character(object) |
Abfrage, ob das Objekt vom Datentyp character ist. Es
wird TRUE oder
FALSE zurückgegeben.
|
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
is.logical(object) |
Abfrage, ob das Objekt vom Datentyp logical ist. Es
wird TRUE oder
FALSE zurückgegeben.
|
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
is.numeric(object) |
Abfrage, ob das Objekt vom Datentyp numeric ist. Es
wird TRUE oder
FALSE zurückgegeben.
|
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
length(object) |
Ermittelt die Anzahl der Elemente eines Objekts. Ein leeres Objekt
(mit Wert NULL) hat die Länge 0, eine Funktion die
Länge 1. |
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
matrix(data = NA, nrow = 1, ncol = 1, byrow = FALSE, dimnames = NULL)
|
Erstellen einer Matrix, die aus den Werten eines Vektors
(data) über
eine festzulegende Anzahl Zeilen (nrow) und Spalten
(ncol) aufgespannt wird. Sie kann entweder zeilen-
(byrow = TRUE)
oder spaltenweise (byrow = FALSE) befüllt werden. Die
Namen der Zeilen und Spalten sind über dimnames in
einer Liste von
Vektoren zu übergeben (beispielsweise
dimnames =
list(c("Zeile1", "Zeile2", "Zeile3"), c("Spalte1", "Spalte2"))).
|
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
mode(object) |
Bestimmung des Datentyps eines Objekts. Mögliche Rückgabewerte:
numeric, character und
logical.
|
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
rbind(...) |
Hinzufügen einer oder mehrerer Zeilen (in Vektorform) zu einer
Matrix (beispielsweise rbind(matrix1, vector1)). Wenn
die
Anzahl der Elemente des Vektors die Anzahl der Elemente in einer
Zeile der Matrix unterschreitet, wird der letzte Wert
des Vektors recycelt. |
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
rownames(object) |
Ausgabe der Zeilennamen eines Objekts, bei dem es sich um eine Matrix oder um einen Dataframe handelt. | ? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
setwd(directory) |
Ändern des Arbeits- beziehungsweise Zielverzeichnisses.
directory
gibt den absoluten Pfad an (beispielsweise
setwd("/home/users/sschneider")).
|
? | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
R-Skripte
Datenbankabfragen mit R und SQL
Das vorliegende, kommentierte Skript dient dazu, über das DHVLab mit R und dem Paket RMySQL eine Verbindung zu einer MySQL-Datenbank herzustellen und auf Basis dieser Datenbank weiterführende Analysen durchzuführen. In den nachfolgenden Abschnitten Abfragen mit SQL und Abfragen mit R werden jeweils dieselben Fragestellungen abgehandelt, um Unterschiede in der Syntax der beiden Sprachen aufzudecken. Der Part mit Abfragen in SQL setzt Grundlagen in SQL voraus, der Part mit Abfragen in R entsprechend Grundlagen in R.
Für die Ausführung der Befehle ist ein freigeschalteter Account des
DHVLab notwendig. Konkret wird der Bestand des Onlinespiels ARTigo betrachtet,
in dem Kunstwerke
verschlagwortet werden. Die Struktur der Datenbank Artigo können Sie
zuvor im Datenbankmanagementsystem phpMyAdmin
einsehen, indem Sie über die Navigation auf der linken Seite
all -> art -> all_art_artigo anwählen (Abbildung
1). Für dieses Skript sind die Tabellen artworks,
tag und tagging relevant.
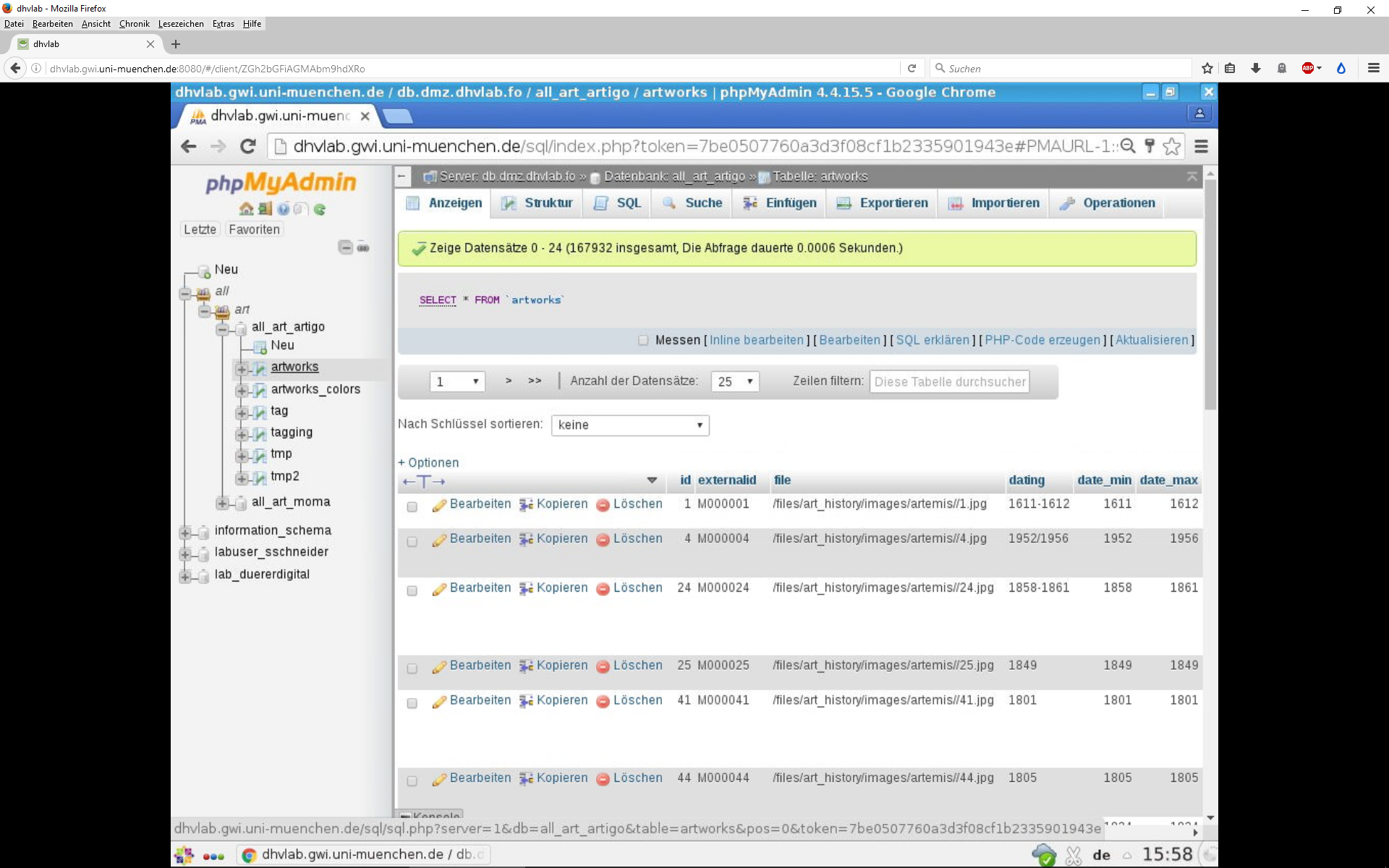
In dieser Einheit verwendete Funktionen: library,
dbConnect, dbGetQuery,
dbListTables, colnames, nrow,
merge, unique, aggregate,
order.
Verbindung zur Datenbank herstellen
# Notwendige Pakete laden
library(RMySQL)
# Mit Datenbank verbinden (Statt "ihrname" und "ihrpasswort" tragen Sie Ihre bei der Registrierung am DHVLab erhaltenen Benutzerdaten ein)
connect <- dbConnect(MySQL(), host = "db.dmz.dhvlab.fo", user = "ihrname", password = "ihrpasswort", dbname = "[Name der Datenbank]")
# Wenn Sie vermeiden möchten, dass Ihr Passwort als plaintext in der Konsole wiedergegeben wird (z.B. im Unterrichtskontext), können Sie alternativ
# eine cnf-Datei (abgelegt in Ihrem persönlichen Verzeichnis auf dem Virtuellen Desktop) mit Ihren Login-Daten für die DB-Verbindung mit R verwenden:
rmysql.settingsfile <- "/home/users/[Benutzername]/.my.cnf"
connect <- dbConnect(MySQL(), default.file = rmysql.settingsfile, dbname = "[Name der Datenbank]")
In der .my.cnf-Datei werden folgende Informationen hinterlegt:
[client]
host = db.dmz.dhvlab.fo
user = [Ihre DHVLab-Nutzerkennung]
password = [Passwort in Plaintext]
# Zu UTF-8 kodieren
dbGetQuery(connect, "set names utf8")
# Tabellen anzeigen
dbListTables(connect)Abfragen mit SQL
# Alle Kunstwerke von Albrecht Dürer
sqlDürer <- dbGetQuery(connect,
"select *
from artworks
where fullname = 'Albrecht Dürer'")
# Anzahl aller Kunstwerke von Albrecht Dürer
sqlDürerAnzahl <- dbGetQuery(connect,
"select count(id) as Anzahl
from artworks
where fullname = 'Albrecht Dürer'")
# Alle Tags, mit denen Werke von Dürer ausgezeichnet worden sind
sqlDürerTag <- dbGetQuery(connect,
"select a.id, a.title, t.tag_id
from artworks a
join tagging t on t.resource_id = a.id
where a.fullname = 'Albrecht Dürer'")
# Alle verschiedenen Tags, mit denen Werke von Dürer ausgezeichnet worden sind
sqlDürerTagDistinct <- dbGetQuery(connect,
"select distinct a.id, a.title, t.tag_id
from artworks a
join tagging t on t.resource_id = a.id
where a.fullname = 'Albrecht Dürer'")
# Alle verschiedenen Tags als Klarname, mit denen Werke von Dürer ausgezeichnet worden sind
sqlDürerTagDistinctName <- dbGetQuery(connect,
"select distinct a.id, a.title, b.name
from artworks a
join tagging t on t.resource_id = a.id
join tag b on b.id = t.tag_id
where a.fullname = 'Albrecht Dürer'")
# Anzahl Tags, die pro Werk vergeben wurden, in absteigender Reihenfolge
sqlDürerTagAnzahl <- dbGetQuery(connect,
"select a.id, a.title, count(*) as Anzahl
from artworks a
join tagging t on t.resource_id = a.id
where a.fullname = 'Albrecht Dürer'
group by a.id
order by Anzahl desc")
# Wie vorhergehende Abfrage, jedoch nur die ersten 10 Resultate zurückgeben
sqlDürerTagAnzahl10 <- dbGetQuery(connect,
"select a.id, a.title, count(*) as Anzahl
from artworks a
join tagging t on t.resource_id = a.id
where a.fullname = 'Albrecht Dürer'
group by a.id
order by Anzahl desc
limit 10")
# Anzahl der verschiedenen Tags pro Werk
sqlDürerTagAnzahlDistinct <- dbGetQuery(connect,
"select a.id, a.title, count(distinct t.tag_id) as Anzahl
from artworks a
join tagging t on t.resource_id = a.id
where a.fullname = 'Albrecht Dürer'
group by a.id
order by Anzahl desc")
# Anzahl der verschiedenen Tags für alle Selbstbildnisse Dürers
sqlDürerTagAnzahlDistinctSelbst <- dbGetQuery(connect,
"select a.id, a.title, count(distinct t.tag_id) as Anzahl
from artworks a
join tagging t on t.resource_id = a.id
where a.fullname = 'Albrecht Dürer' and a.title like 'Selbst%'
group by a.id
order by Anzahl desc")Abfragen mit R
# Alle Daten von artworks, tagging und tag einlesen
artworks <- dbGetQuery(connect, "select * from artworks")
tagging <- dbGetQuery(connect, "select * from tagging")
tag <- dbGetQuery(connect, "select * from tag")
# Abfrage aller Spaltennamen
colnames(artworks)
colnames(tagging)
colnames(tag)
# Alle Kunstwerke von Albrecht Dürer
rDürer <- artworks[which(artworks$fullname == "Albrecht Dürer"),]
# Anzahl aller Kunstwerke von Albrecht Dürer
nrow(rDürer)
# Alle Tags, mit denen Werke von Dürer ausgezeichnet worden sind
rDürerTag <- merge(rDürer, tagging, by.x = "id", by.y = "resource_id") # Tabellen verknüpfen
rDürerTag <- rDürerTag[,c("id","title","tag_id")] # Spalten auswählen
# Alle verschiedenen Tags, mit denen Werke von Dürer ausgezeichnet worden sind
rDürerTagDistinct <- unique(rDürerTag)
# Alle verschiedenen Tags als Klarname, mit denen Werke von Dürer ausgezeichnet worden sind
rDürerTagDistinctName <- merge(rDürerTagDistinct, tag[,c("id","name")], by.x = "tag_id", by.y = "id")
# Anzahl Tags, die pro Werk vergeben wurden, in absteigender Reihenfolge
rDürerTagAnzahl <- aggregate(tag_id ~ id + title, rDürerTag, length)
rDürerTagAnzahl <- rDürerTagAnzahl[order(rDürerTagAnzahl$tag_id, decreasing = TRUE),]
# Wie vorhergehende Abfrage, jedoch nur die ersten 10 Resultate zurückgeben
rDürerTagAnzahl10 <- rDürerTagAnzahl[1:10,]
# Anzahl der verschiedenen Tags pro Werk
rDürerTagAnzahlDistinct <- aggregate(tag_id ~ id + title, rDürerTag, function(x){length(unique(x))})
rDürerTagAnzahlDistinct <- rDürerTagAnzahlDistinct[order(rDürerTagAnzahlDistinct$tag_id, decreasing = TRUE),]
# Anzahl der verschiedenen Tags pro Selbstbildnis
rDürerTagAnzahlDistinctSelbst <- rDürerTagAnzahlDistinct[grepl("Selbst", rDürerTagAnzahlDistinct$title),]Verbindung zur Datenbank schließen
on.exit(dbDisconnect(connect))Cluster und Korrelationen
Das vorliegende, kommentierte Skript dient dazu, über das DHVLab mit R und dem Datensatz Artworks des Museum of Modern Art Analysen zum Themenkreis Cluster und Korrelationen durchzuführen.
In dieser Einheit verwendete Funktionen: read.csv,
gsub, as.numeric, plot,
abline, points, hist,
boxplot, shapiro.test, cor,
quantile, IQR.
Korrelationen
# Daten einlesen
url <- "https://media.githubusercontent.com/media/MuseumofModernArt/collection/master/Artworks.csv"
artworks <- read.csv(url)
# Übersicht über die Daten
colnames(artworks)
table(as.factor(artworks$Classification))
# Daten selektieren
paintings <- artworks[artworks$Classification == "Painting",]
paintings <- paintings[,c("Title", "Artist", "Date", "DateAcquired", "Height..cm.", "Width..cm.")]
#------------------------------------------------
# Zusammenhang Entstehungsdatum und Ankaufdatum
paintings$Date <- gsub(".*?([1-2][0-9]{3}).*", "\\1", paintings$Date)
paintings$Date <- as.numeric(paintings$Date)
paintings$DateAcquired <- gsub(".*?([1-2][0-9]{3}).*", "\\1", paintings$DateAcquired)
paintings$DateAcquired <- as.numeric(paintings$DateAcquired)
paintingsDate <- paintings[!is.na(paintings$Date) & !is.na(paintings$DateAcquired),]
# Streudiagramm
plot(paintingsDate$Date, paintingsDate$DateAcquired, main = "Zusammenhang Entstehungsdatum und Ankaufdatum", xlab = "Entstehungsdatum", ylab = "Ankaufdatum")
# Verteilungen der Variablen
hist(paintingsDate$Date)
boxplot(paintingsDate$Date)
shapiro.test(paintingsDate$Date)
hist(paintingsDate$DateAcquired)
boxplot(paintingsDate$DateAcquired)
shapiro.test(paintingsDate$DateAcquired)
# Korrelation mit Kendalls Tau (Moderater Zusammenhang), da metrische, nicht normalverteilte Variablen
cor(paintingsDate$Date, paintingsDate$DateAcquired, method = "kendall")
#------------------------------------------------
# Zusammenhang Höhe und Breite
paintingsDim <- paintings[!is.na(paintings$Height..cm.) & !is.na(paintings$Width..cm.),]
# Streudiagramm
plot(paintingsDim$Width..cm., paintingsDim$Height..cm., main = "Zusammenhang Breite und Höhe", xlab = "Breite", ylab = "Höhe")
abline(lm(paintingsDim$Height..cm. ~ paintingsDim$Width..cm.), col = "red")
# Ausreißer kennzeichnen
qWidth <- quantile(paintingsDim$Width..cm., 0.75)
iqrWidth <- IQR(paintingsDim$Width..cm.)*1.5
qHeight <- quantile(paintingsDim$Height..cm., 0.75)
iqrHeight <- IQR(paintingsDim$Height..cm.)*1.5
outliers <- paintingsDim[paintingsDim$Width..cm. > (qWidth + iqrWidth) | paintingsDim$Height..cm. > (qHeight + iqrHeight),]
points(outliers$Width..cm., outliers$Height..cm., col = "green")
# Verteilungen der Variablen
hist(paintingsDim$Width..cm.)
boxplot(paintingsDim$Width..cm.)
shapiro.test(paintingsDim$Width..cm.)
hist(paintingsDim$Height..cm.)
boxplot(paintingsDim$Height..cm.)
shapiro.test(paintingsDim$Height..cm.)
# Korrelation mit Kendalls Tau (Starker Zusammenhang), da metrische, nicht normalverteilte Variablen
cor(paintingsDim$Width..cm., paintingsDim$Height..cm., method = "kendall")Cluster
Coming Soon.